Projekte
Der Erreger der Lungentuberkulose beim Menschen, Mycobacterium tuberculosis, ist das bedeutsamste bakterielle Pathogen. Trotz jahrzehntelanger Forschung sterben weiterhin jährlich 1,5 Millionen Menschen an dieser armutsassoziierten Infektionskrankheit. Trotz der langen zurückliegenden Entschlüsselung des Genoms von Mycobacterium tuberculosis in 1998 ist die Funktion und Relevanz einer Vielzahl an Genen immer noch nicht aufgeklärt worden. Um einen Beitrag zur Bekämpfung der globalen Tuberkulosepandemie zu liefern, wendet die Arbeitsgruppe Kalscheuer eine umfassende molekulargenetische Manipulierung von Mycobacterium tuberculosis an, um die Bedeutung von Genen unbekannter Funktion aufzuklären. Unter Verwendung von spezialisierter Transduktion mittels temperatur-sensitiven Mycobacteriophagen stellen wir Gendeletionsmutanten, konditionale Mutanten, merodiploide Stämme und Überexpressionsstämme her, um spezifische Fragestellungen adressieren zu können. Hierüber sollen essentielle Gene und Virulenzfaktoren identifiziert und anschließend umfassend molekularbiologisch und biochemisch hinsichtlich ihrer biologischen Funktion charakterisiert werden. Die untersuchten Genprodukte können potentielle neue Antibiotikawirkorte darstellen und ein rationales strukturbasiertes Design von Wirkstoffen erlauben. Insbesondere ist die Arbeitsgruppe an Genen mit einer Rolle in der Zellwandbildung und in der Biosynthese von Glycokonjugaten in Mycobacterium tuberculosis interessiert.

Ausgewählte Publikationen:
Babu Sait MR, Koliwer Brandl H, Stewart JA, Swarts BM, Jacobsen M, Ioerger TR, Kalscheuer R. PPE51 mediates uptake of trehalose across the mycomembrane of Mycobacterium tuberculosis. Sci Rep 12, 2097 (2022).
Kamariza M, Shieh P, Ealand CS, Peters JS, Chu B, Rodriguez-Rivera FP, Babu Sait MR, Treuren WV, Martinson N, Kalscheuer R, Kana BD, Bertozzi CR. Rapid detection of Mycobacterium tuberculosis in sputum with a solvatochromic trehalose probe. Sci Translat Med 10, eaam6310 (2018).
Das pharmakologische Potenzial von Naturstoffen ist nicht erst seit der Entdeckung des Penicillins und seiner antibakteriellen Eigenschaften im Jahre 1928 bekannt. Gerade aus Bakterien und mikrobiellen Pilzen wurde seither eine Vielzahl, heute aus medizinischer Sicht nicht mehr wegzudenkender Verbindungen isoliert. Die Entdeckung neuer und bekannter Naturstoffe mikrobiellen Ursprungs mit Fokus auf unbeschriebene antimikrobielle Bioaktivitäten ist deshalb eines unserer grundlegenden Ziele, um an der immer dringender werdenden Bekämpfung einer sich zuspitzenden mikrobiellen Resistenzlage mitzuwirken.
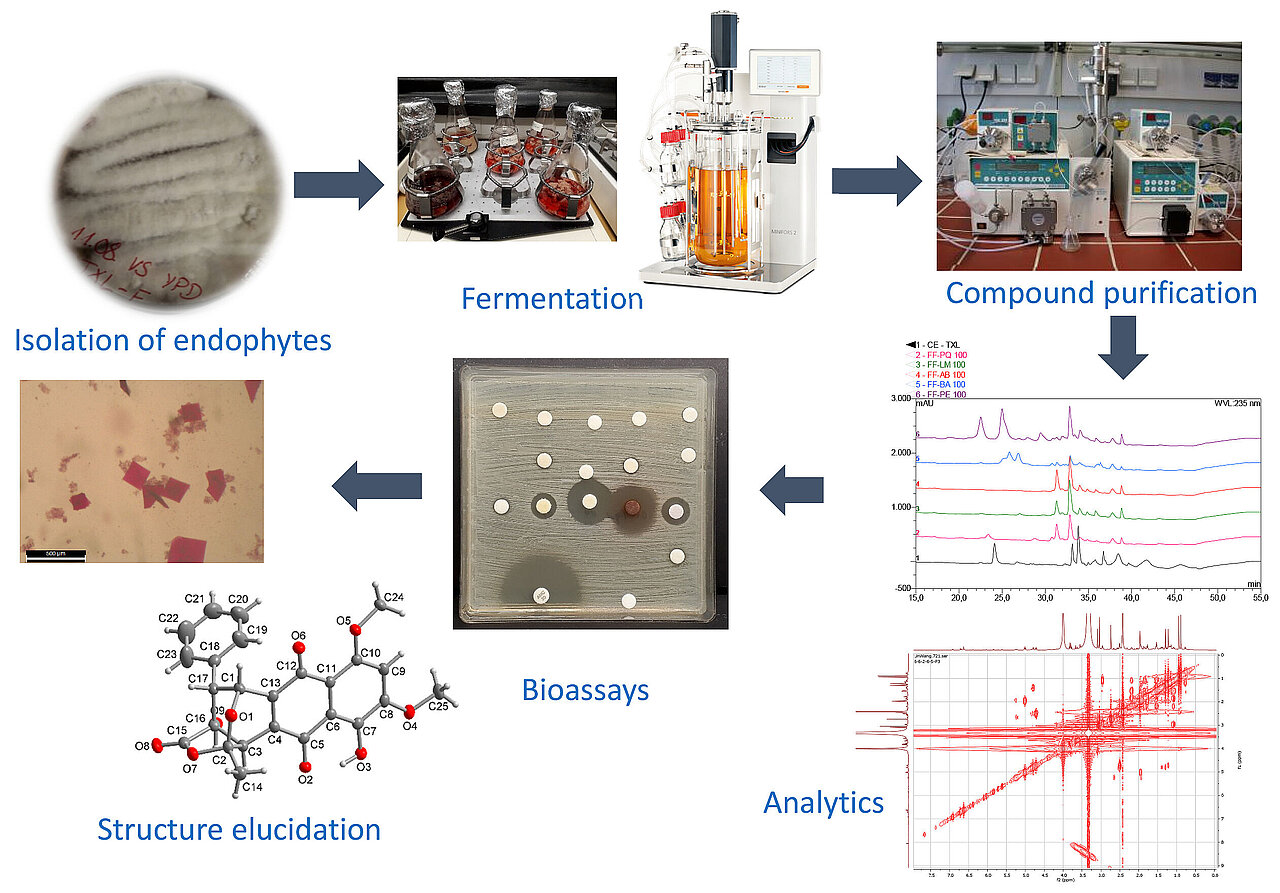
Das Arbeitsspektrum beginnt mit der Isolation von Mikroorganismen aus diversen Umweltproben oder von endophytischen Pilzen und Bakterien aus Pflanzen(teilen). Lösungsmittelextrakte, die aus Biomasse und Kulturüberständen dieser Isolate gewonnen werden, werden dann auf antimikrobielle Aktivitäten gegen ein breites Spektrum humanpathogener Mikroorganismen getestet. Die Aufreinigung der Rohextrakte bis hin zu den isolierten reinen Substanzen erfolgt Bioaktivitäts-geleitet über verschiedene präparative Methoden wie Flüssig-Flüssig-Extraktion, VLC (vacuum liquid chromatography), Sephadex-Chromatographie, RP-VLC (reverse-phase vacuum liquid chromatography) oder semipräparative HPLC. Die erhaltenen Reinstoffe werden anschließend über verschiedene analytische Methoden, wie MS (mass spectrometry), ein- und zweidimensionales NMR (nuclear magnetic resonance) und die Aufnahme eines UV-Vis-Spektrums strukturell aufgeklärt. Eine nähergehende stereochemische Aufklärung kann über spezielle NMR-Verfahren, Polarimetrie, chemische Derivatisierungsreaktionen, röntgenkristallographische Aufnahmen und die Messung des Circulardichroismus erfolgen. Neue aktive Verbindungen und bekannte Verbindungen mit unbeschriebener Aktivität werden anschließend in nähergehenden antimikrobiellen und zytotoxischen Assays auf ihr antimikrobielles und zelltoxisches Potenzial hin untersucht und charakterisiert.
Ausgewählte Publikationen:
Eze PM, Simons V, Seidemann T, Wang L, Kiffe-Delf AL, Frank M, van Geelen L, Abba CC, Esimone CO, Okoye FBC, Kalscheuer R. Serratiochelins A and B from Serratia marcescens show xenosiderophoric characteristics towards Acinetobacter baumannii and Mycobacterium tuberculosis. Trop J Pharm Res 20, 2551-2658 (2021).
Eze P, Gao Y, Liu Y, van Geelen L, Ejikeugwu C, Esimone C, Okoye F, Proksch P, Kalscheuer R. Secondary metabolites of a marine-derived Penicillium ochrochloron. Not Sci Biol 13, 11020 (2021).
Die rasante Zunahme antimikrobieller Resistenz ist ein weltweit zunehmendes Problem und wird auch als Antimikrobielle Resistenz Krise (AMR-Krise) bezeichnet. Dies ist nicht nur eine Belastung für die medizinische Versorgung, sondern stellt viele Länder auch vor ökonomische Herausforderungen. Die Weltgesundheitsorganisation (WHO) hat vor einigen Jahren reagiert und eine Liste von Pathogenen erstellt, für die es bereits jetzt oder in naher Zukunft nur noch wenige effektive Therapien geben wird, wenn sich der Gebrauch von Antibiotika nicht drastisch ändert und die Verbreitung von Resistenzen im derzeitigen Tempo weitergeht. Diese Bakterien werden unter dem Apronym ESKAPE zusammengefasst und umfassen sowohl Gram-positive (Enterococcus faecium und Staphylococcus aureus) als auch Gram-negative humanpathogene Bakterien (Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa und die Enterobacter spp.).
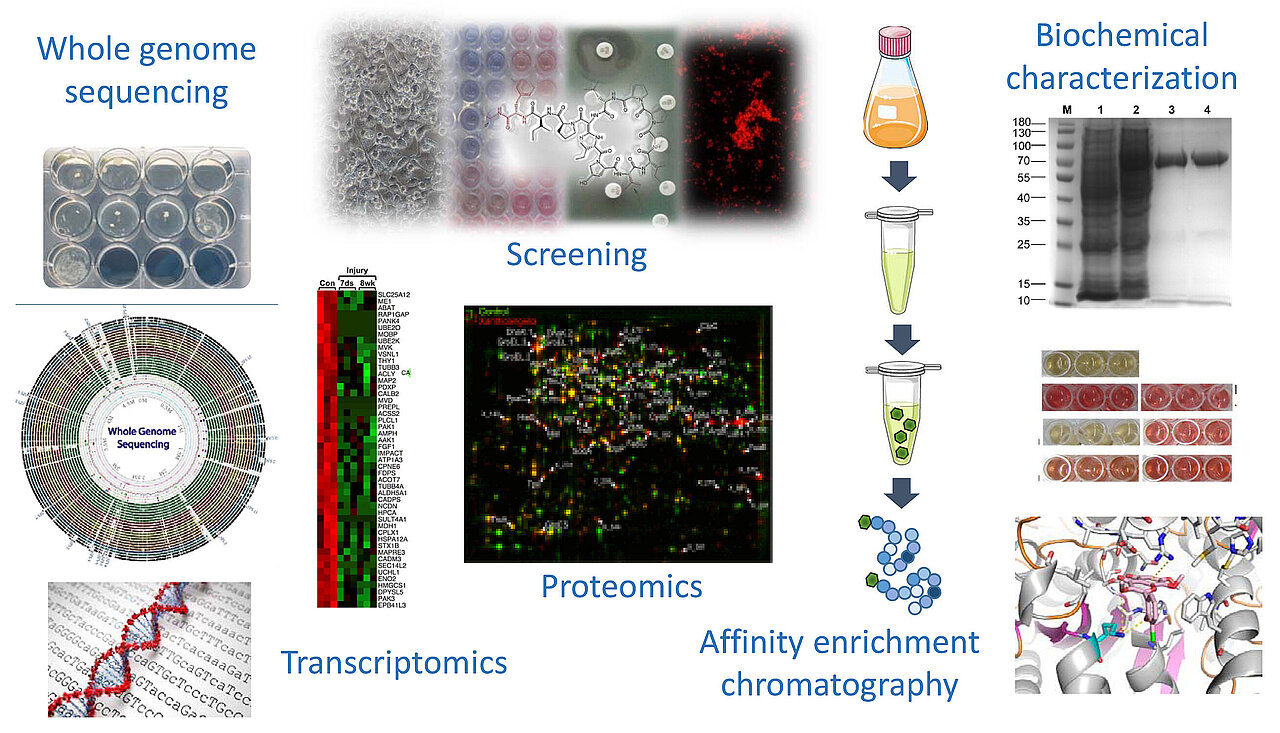
Unsere Arbeit beginnt mit dem Screening sowohl von isolierten Naturstoffen als auch von (semi-)synthetischen Molekülen auf ihr antimikrobielles Potential. Es werden neben den ESKAPE Pathogenen auch Pilze wie Candida albicans sowie Mycobacterium tuberculosis, der Erreger der Lungentuberkulose, in das Screening eingeschlossen. Verschiedene humane Zelllinien werden für das Testen auf zytotoxische Eigenschaften verwendet, um antimikrobielle Moleküle mit adäquater therapeutischer Breite zu identifzieren. Methoden wie u.a. die Generierung spontan resistenter Mutanten, Genomsequenzierung, Abtötungskinetiken, Transkriptom- und Proteomanalysen von Stressantworten von wirkstoffbehandelten Zellen als auch verschiedenste biochemische und gentechnische Methoden werden verwendet, um die antimikrobiellen Eigenschaften der aussichtsreichsten Moleküle weiter zu charakterisieren und den Wirk- und Resistenzmechanismus aufzuklären. Diese Arbeiten sollen dazu beitragen, der AMR-Krise entgegenzuwirken und neue effektive Therapeutika für die Behandlung von Infektionen mit multi-resistenten Pathogenen zu entwickeln.
Ausgewählte Publikationen:
Rehberg N, Sommer G, Drießen D, Kruppa M, Adeniyi ET, Chen S, Wang L, Wolf K, Tasch B, Ioerger TR, Zhu K, Müller TJJ, Kalscheuer R. Nature-inspired (di)azine-bridged bisindole alkaloids with potent antibacterial in vitro and in vivo efficacy against Methicillin-resistant Staphylococcus aureus. J Med Chem 63, 12623-12641 (2020).
van Geelen L, Kaschani F, Sazzadeh SS, Adeniyi ET, Meier D, Proksch P, Pfeffer K, Kaiser M, Ioerger TR, Kalscheuer R. Natural brominated phenoxyphenols kill persistent and biofilm-incorporated cells of MRSA and other pathogenic bacteria. Appl Microbiol Biotechnol 104, 5985-5998 (2020).
Meier D, Vázquez Hernández M, van Geelen L, Muharini R, Proksch P, Bandow JE, Kalscheuer R. The plant-derived chalcone Xanthoangelol targets the membrane of Gram-positive bacteria. Bioorg Med Chem 27, 115151 (2019).
Rehberg N, Omeje E, Ebada SS, van Geelen L, Liu Z, Sureechatchayan P, Kassack MU, Ioerger TR, Proksch P, Kalscheuer R. 3-O-Methyl-alkylgallates inhibit fatty acid desaturation in Mycobacterium tuberculosis. Antimicrob Agents Chemother 63, e00136-19 (2019).
Rehberg N, Akone HS, Ioerger TR, Erlenkamp G, Daletos G, Gohlke H, Proksch P, Kalscheuer R. Chlorflavonin targets acetohydroxyacid synthase catalytic subunit IlvB1 for synergistic killing of Mycobacterium tuberculosis. ACS Infect Dis. 4, 123-134 (2018).

